Abstrakt
Der größte Primat aller Zeiten und eine der größten der südostasiatischen Megafauna, Gigantopithecus blacki1, überlebte in China etwa 2,0 Millionen Jahre lang bis zum späten Mittelpleistozän, als er ausstarb2,3,4. Sein Untergang ist rätselhaft, wenn man bedenkt, daß er einer der wenigen asiatischen Menschenaffen war, die in den letzten 2,6 Millionen Jahren ausgestorben sind, während andere, darunter der Orang-Utan, bis heute überlebt haben5. Die Ursache für das Verschwinden von G. blacki bleibt ungeklärt, könnte aber Aufschluß über die Widerstandsfähigkeit der Primaten und das Schicksal der Megafauna in dieser Region geben6. Hier haben die Forscher drei multidisziplinäre Analysen – Zeitablauf, vergangene Umgebungen und Verhalten – auf 22 Höhlen in Südchina angewendet. Sie haben 157 radiometrische Alter aus sechs Datierungstechniken verwendet, um einen Zeitplan für das Ableben von G. blacki zu erstellen. Sie zeigen, daß die Umwelt vor 2,3 Millionen Jahren ein Mosaik aus Wäldern und Gräsern war, das ideale Bedingungen für gedeihende G. blacki-Populationen bot. Kurz vor und während des Aussterbefensters vor 295.000 bis 215.000 Jahren kam es jedoch aufgrund der zunehmenden Saisonalität zu einer erhöhten Umweltvariabilität, die zu Veränderungen in den Pflanzengemeinschaften und einer Zunahme offener Waldumgebungen führte. Obwohl es seinem nahen Verwandten Pongo weidenreichi gelang, seine Ernährungspräferenzen und sein Verhalten an diese Variabilität anzupassen, zeigte G. blacki Anzeichen von chronischem Stress und schrumpfenden Populationen. Letztendlich führte sein Anpassungskampf zum Aussterben des größten Primaten, der jemals auf der Erde gelebt hat.
Kerninhalt
Unser derzeitiges Verständnis von Gigantopithecus blacki beruht auf Höhlenablagerungen des frühen bis mittleren Pleistozäns in Südchina zwischen dem Jangtsekiang und dem Südchinesischen Meer (Abb. 1 und Abschnitt 1 mit ergänzenden Informationen). Diese Pongine7 gilt als Schlüsselmitglied der früh- bis mittelpleistozänen Faunenzonen Gigantopithecus–Sinomastodon und Stegodon–Ailuropoda im (sub)tropischen Ostasien, von etwa 2,0 Millionen Jahren (Ma) bis 330 Tausend Jahren (ka) 2,3 ,8,9. Er ist bekannt für seine ungewöhnlich großen Backenzähne, seine atypische Schmelzdicke, seine geschätzte Körpergröße von etwa 3 m und sein Gewicht von 200–300 kg und ist damit der größte Primat, der jemals auf der Erde gelebt hat4. Trotz 85 jähriger Suche beschränkt sich der Fossilienbestand von G. blacki auf vier Mandibeln und fast 2.000 isolierte Zähne ohne postkranielle Beweise4. Seine erste Entdeckung in einer Apotheke in Hongkong als „Drachenzahn“1 löste eine Suche nach den ersten In-situ-Funden aus10 (Weiterführende Abbildung 1f) und gipfelte in der Entdeckung mehrerer Höhlenstandorte in zwei Hauptgebieten, Chongzuo und Bubing Becken, in der ZAR-Provinz Guangxi4. Diese Stätten enthalten entscheidende Beweise für sein Überleben und seinen späteren Untergang.
Fig. 1: Die Verortung der Studiengebiete dieser Forschungsergebnisse.
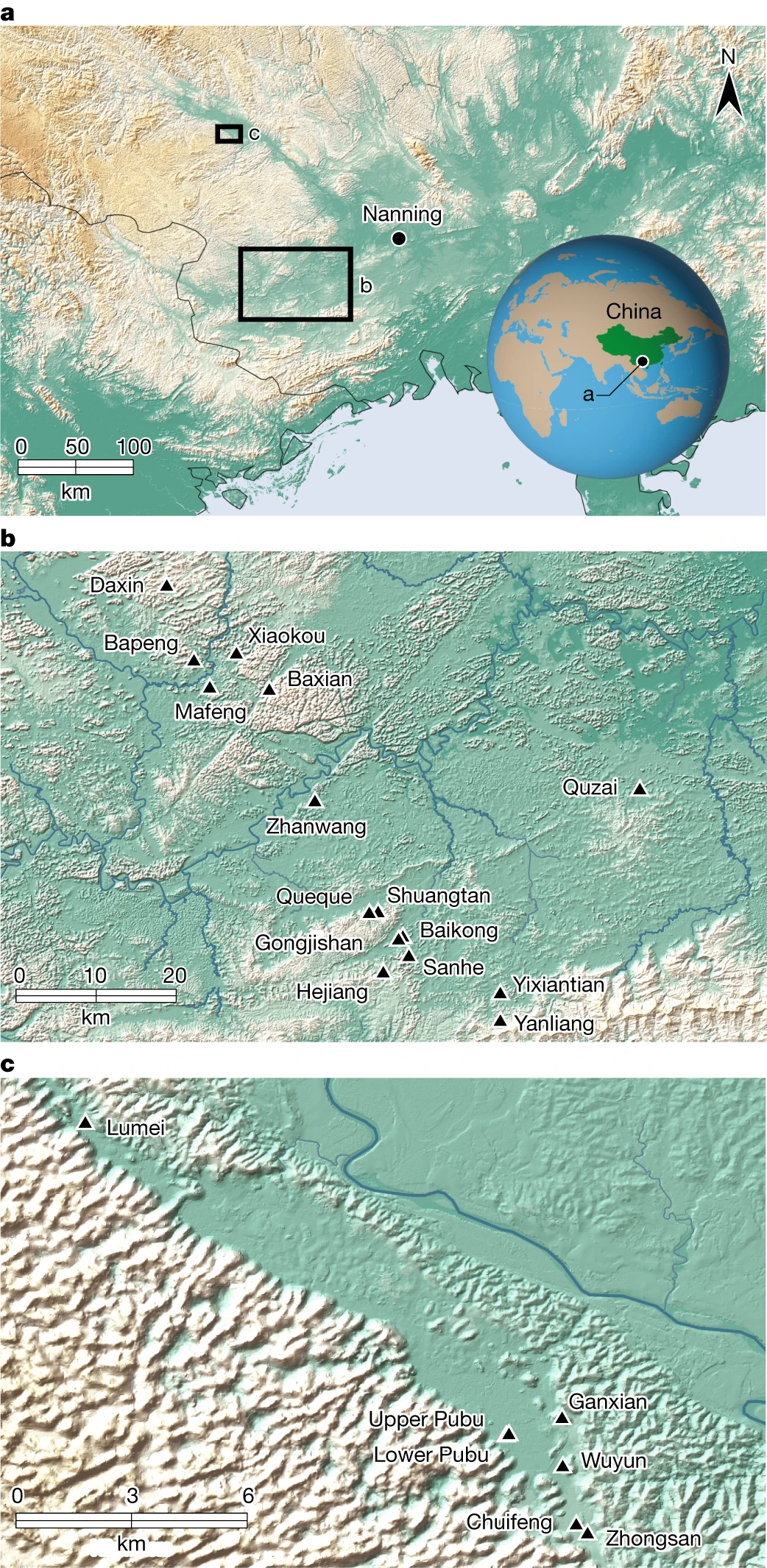
a–c, Die Lage von Südchina, der ZAR-Provinz Guangxi und der Stadt Nanning (a), wobei die Lage des Chongzuo-Untersuchungsgebiets durch ein großes Kästchen (b) und das Bubing-Becken-Untersuchungsgebiet durch ein kleineres Kästchen markiert ist ( C). b, Die Lage der 16 analysierten Höhlenstandorte im Chongzuo-Untersuchungsgebiet. c, Die Lage der sechs Höhlen, die im Untersuchungsgebiet des Bubing-Beckens analysiert wurden, einschließlich sowohl G. blacki-tragender als auch nicht-G. blacki-haltiger Höhlen. Blacki-haltige Höhlen aus beiden Regionen.
Nur sehr wenige dieser G. blacki-Standorte wurden mit mehr als einer radiometrischen Technik datiert; Daher bleibt der Zeitpunkt des Aussterbens ungewiss11. Die aktuelle Zeitspanne für sein Vorkommen liegt zwischen 2,2 Ma (Baikong-Höhle12) und 420–330 ka (Hejiang-Höhle9). Während dieser Zeit erfuhr G. blacki morphologische Veränderungen, einschließlich einer Zunahme der Zahngröße13 und der Zahnkomplexität9, was offenbar auf eine Ernährungsumstellung als Reaktion auf den ökologischen Druck hindeutet13. Rekonstruktionen der Ernährung von G. blacki auf der Grundlage der Zahnanatomie weisen auf einen spezialisierten Pflanzenfresser mit Anpassungen für den Verzehr abrasiver Nahrung14,15, starkes Kauen faseriger Nahrung16,17 und eine fruchtreiche Ernährung6,18 hin. Das vielfältige Waldökosystem zur Zeit von Baikong war in der Lage, die Biomasse mehrerer Primatengemeinschaften4 in einem weiten Gebiet in den Provinzen Guangxi, Guizhou, Hainan und Hubei zu unterstützen19. Zur Zeit von Hejiang war das Verbreitungsgebiet von G. blacki jedoch drastisch auf nur noch Guangxi9,13 zurückgegangen. Die Gründe für diesen dramatischen Rückgang und das letztendliche Aussterben sind nach wie vor heftig umstritten4, da es an einem regionalen Ansatz, einer Konzentration auf einzelne Standorte und Methoden und dem Fehlen von Verhaltens-4 und Umweltbeweisen20 mangelt.
Um die möglichen Ursachen für das Aussterben von G. blacki zu identifizieren, haben die Wissenschaftler einen regionalen Ansatz auf 22 Höhlen in Chongzuo und Bubing Basin angewendet, die entweder G. blacki-tragend (11) oder nicht-G. blacki-haltig waren. Blacki-haltige (11) Höhlenablagerungen (Erweiterte Daten, Abb. 1 und 2 und Ergänzende Informationen, Abschnitte 2 und 3). Mithilfe einer Kombination aus früheren Ausgrabungen (1999–2016) und neu entdeckten Höhlen (2017–2020) identifizierten und beprobten die Forscher fossile Brekzien für Datierungen, Paläoklima-Proxies und Verhaltensanalysen. Sie haben sechs unabhängige Datierungstechniken auf die Sedimente (Post-Infrarot-Infrarot-stimulierte Lumineszenz (pIR-IRSL), optisch stimulierte Lumineszenz (OSL), Elektronenspinresonanz (ESR) auf Quarz und U-Serie auf Speläothem) und Fossilien (U-Infrarot-stimulierte Lumineszenz) angewendet. Reihen zu Zähnen, gekoppelt US-ESR), um einen Bayes’schen modellierten Altersbereich für jeden Standort zu bestimmen (Ergänzende Informationen, Abschnitte 4–8), die dann weiter modelliert wurden, um ein regionales Aussterbefenster (EW) bereitzustellen. Sie haben Pollen-, Holzkohle-, paläontologische, stabile Isotopen- und mikrostratigraphische Analysen auf die Sedimente und Fossilien angewendet, um die früheren Umgebungen zu rekonstruieren (Ergänzende Informationen, Abschnitte 3 und 10–12). Schließlich haben wir eine Spurenelement-, stabile Isotopen- und Dental-Microwear-Texturanalyse (DMTA) auf die Zähne von G. blacki und den engsten Verwandten von Pongo weidenreichi angewendet, um etwaige Veränderungen in der Ernährung und im Verhalten von G. blacki vor und innerhalb der EW zu bestimmen, die damit in Zusammenhang stehen könnten bis zu seinem Untergang (Ergänzende Informationen, Abschnitte 12–14).
Den 157 radiometrischen Altersschätzungen zufolge liegen die Fossilfunde in den 22 Höhlen zwischen 2.300 und 49 ka (Abb. 2a und 3a, Erweiterte Daten Abb. 3–6 und Ergänzende Informationen in den Abschnitten 4–8 für alle Datierungstabellen und Diskussion von Einschränkungen). und Unsicherheiten). Diese Studie erweitert die Zeitachse für das Vorkommen von G. blacki von 2,3 Ma auf 255 ka, liefert einen genauen Zeitpunkt für das Fenster des Aussterbens bei 295–215 ka (2σ) (Ergänzende Informationen, Abschnitt 9) und legt Schwerpunkte für die Paläoumwelt und fest Verhaltensanalyse (Prä-EW (2.300–700 ka), Übergangsphase (700–295 ka), EW (295–215 ka) und Post-EW (215 ka bis heute)).
Fig. 2: Beispieldatensätze zur Unterstützung der Aussterbeereignisse.
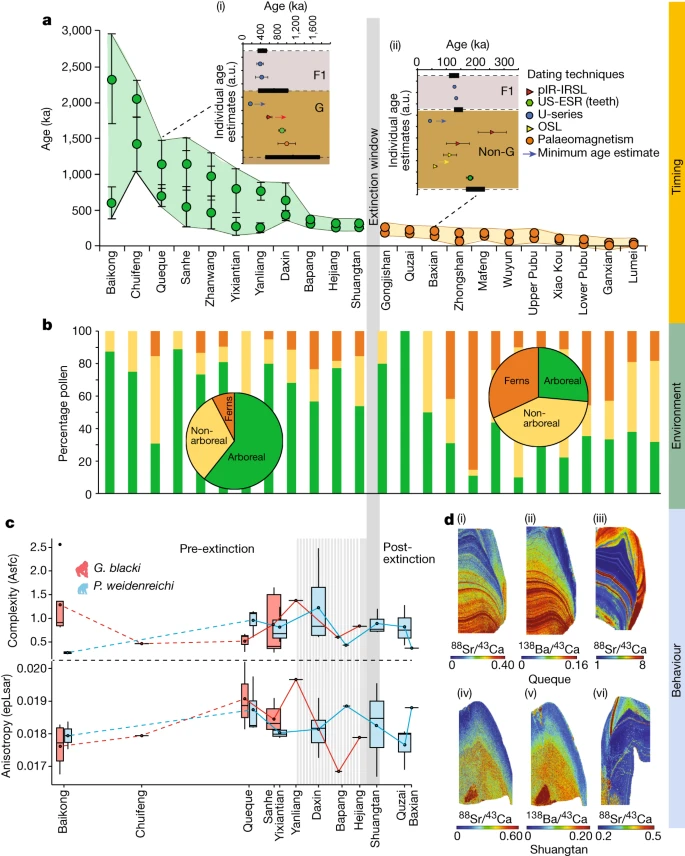
a–d, Daten beziehen sich auf das Timing (a), die Umgebung (b) und das Verhalten (c,d), die von den Standorten präsentiert werden. a, Modellierte Altersbereiche jeder Höhle (n = 22 Höhlen) unter Verwendung des minimalen und maximalen Alters der fossilführenden Einheit (n = 157 Proben). Die Höhlen (x-Achse) im Vergleich zum Alter (y-Achse), mit G. blacki (grüne Kreise) und Nicht-G. Blacki (orangefarbene Kreise) Brekzie. Die Datenpunkte stellen Durchschnittsalter mit Standardabweichung dar. bei 2σ Unsicherheiten. Die Einschübe sind modellierte Brekzien aus Queque (i) und Baxian (ii). G, G. blacki-führende Brekzie; F1, darüber liegender Fließstein; und Non-G, Abwesenheit von G. blacki. Datenpunkte sind Durchschnittsalter mit Standardabweichung. bei 2σ Unsicherheiten. Die schwarzen horizontalen Rechtecke (mit gestrichelten Linien) stellen die Grenze gemäß der Modellierung dar (Ergänzende Informationen, Abschnitt 2 und Ergänzende Abbildung S1a – v). Die modellierte EW ist die vertikale graue Linie. b, Prozentsatz der Pollen von den Standorten in a, die Baumarten (grün), Nichtbaumarten (gelb) und Farne (orange) repräsentieren. Die Kreisdiagramme liefern einen Durchschnitt der Pollenveränderungen vor (links) und nach dem Aussterben (rechts). c, DMTA-Boxplot-Reihe nach Alter von 12 Höhlen (x-Achse) versus molarer Mikroverschleißkomplexität (Asfc, oben, y-Achse) und Anisotropie (epLsar, unten, y-Achse) von G. blacki (rot, n = 16) und P . weidenreichi (blau, n = 22). Die Größenbereiche der Boxplots stellen mittlere Komplexitäts- und Anisotropiewerte pro Standort dar. Die Daten werden als Mittelwerte ± Interquartilbereich und Whiskers bei 95 % KI dargestellt (Ergänzungstabelle S28). d, Spurenelementkartierung von G. blacki und P. weidenreichi. Sr/Ca (i) und Ba/Ca (ii) eines rechten M3 G. blacki-Zahns (CSQSN-44) und Sr/Ca-Karte eines rechten M2 P. weidenreichi-Zahns (CSQ0811-4) (iii), alle aus Queque Höhle. Unten: Sr/Ca (iv) und Ba/Ca (v) aus einem P4-Zahn von G. blacki (ST_02_109) im Vergleich zu Sr/Ca (vi) aus einem linken M3-Zahn von P. weidenreichi (CLMST0911-118), alle aus Shuangtan-Höhle. a.u., beliebige Einheiten.
Abb. 3: Eine Zusammenfassung aller Datensätze, chronologisch aufbereitet.
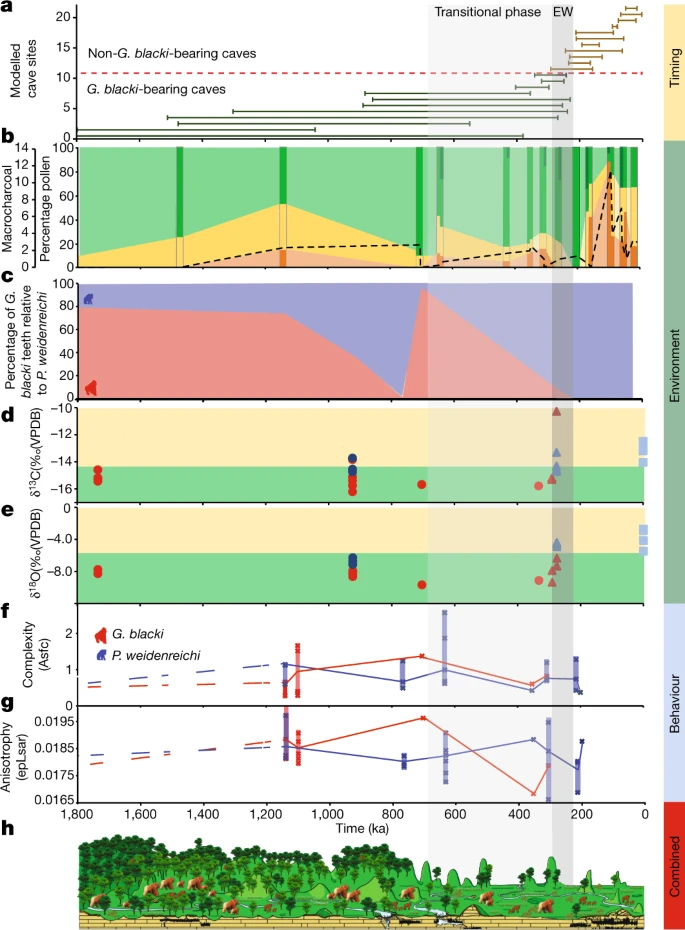
a, Zeitleiste des Aussterbens basierend auf den modellierten Altersspannen für alle 22 Höhlen. Die Zahlen auf der y-Achse beziehen sich auf die Höhlen in Abb. 2a. Beachten Sie die verkürzte Zeitleiste (1.800 ka). Die EW (255 ± 40 ka) ist eine vertikale graue Box (EW) mit einer durchgezogenen, hellgrauen Box (Übergangsphase) für den Beginn einer erhöhten Umweltvariabilität. b, Der Prozentsatz an Pollen, aufgetragen auf einer Zeitachse, gruppiert in baumartige (grün), nicht baumartige (gelb) und Farne (orange). Die dunkleren Streifen stellen Standorte dar, die Pollendaten enthalten, während die helleren Abschnitte dazwischen eine Schätzung der Pollenveränderungen darstellen. Die Mikrokohle (schwarze gestrichelte Linie) korreliert mit der Zunahme der Farne und dem Rückgang der Baumbedeckung. Die dunkelgrünen Baumabschnitte stellen Waldstörungen dar. Taxa mit hohem Umsatz wie Trema, Celtis und Sapindaceae sind während der Übergangsphase und in Ostwesteuropa vorhanden. c, Der Prozentsatz der Zähne von G. blacki (rot) im Verhältnis zu den Zähnen von P. weidenreichi (blau) in repräsentativen Höhlen als grober Indikator für die relative Häufigkeit von G. blacki im Vergleich zu P. weidenreichi an jedem Standort. Die relative Anzahl der G. blacki-Zähne nimmt unmittelbar vor der Übergangsphase ab, was eine Veränderung in der Zusammensetzung der Fauna darstellt, und während der Übergangsphase, was die Ausrottung von G. blacki bedeutet. d,e, Isotopenänderungen für fossile Zähne von P. weidenreichi (blaue Kreise und Dreiecke) und G blacki (rote Kreise und Dreiecke), aufgezeichnet auf einer Zeitachse; moderne P. weidenreichi sind blaue Quadrate. δ13C (‰) (d) und δ18O(‰) (e). f,g, DMTA-Boxplot-Zeitreihen für Mikroverschleißkomplexität (f) und Anisotropie (g) von G. blacki (rot) und P. weidenreichi (blau); Definitionen siehe Abb. 2c. h, Ein Landschafts- und Umweltzeitschnitt, der die Veränderung der Vegetation und der Primatenarten von der Zeit vor der EW über die EW bis zur Zeit nach der EW zeigt.
Dir durchgeführte Pollenanalyse zeigt, daß die Umwelt während der Vor-EW von Baumarten (Pinaceae, Fagaceae und Betulaceae) mit Grünlandflächen dominiert wurde (Abb. 2b und 3b). Allerdings kam es vor der EW während der Übergangsphase zu einer Veränderung der Waldpflanzengemeinschaften und einer Zunahme der Waldstörungstaxa, wobei offenere Wälder dominierten. Nach der EW etwa 200 ka gab es einen starken Rückgang der Baumbedeckung, eine Zunahme der Farne (z. B. Moraceae und Podocarpus), eine starke Zunahme des Graslandes (z. B. Poaceae) und vermehrte Anzeichen von Holzkohle in der Landschaft (Erweitert). Daten Abb. 7 und ergänzende Informationen Abschnitt 10).
Eine detaillierte Faunenanalyse zeigt, daß die Standorte vor der EW durch G. blacki (in relativ großer Zahl) (Abb. 3c), Ailuropoda microta, Procynocephalus, Sinomastodon, Stegodon, Hesperotherium und Hippopotamodon gekennzeichnet waren, die sich zu G. blacki (in relativ großer Zahl) verlagerten kleine Zahlen) (Abb. 3c), Ailuropoda baconi, Stegodon und Elephas vor dem EW und ein Fehlen von G. blacki nach dem EW (Ergänzende Informationen, Abschnitt 3). Die mikrostratigraphischen Analysen von fünf Höhlen zeigen vor-EW-Mikrofazies, die von feinen Körnern, höheren Tonen und Oxiden, Bioturbation und Guano-induzierter Phosphatierung dominiert werden. Im EW nahmen die Korngrößen zu, wobei geringere Oxide, Bioturbation und Knochen-/Zahnveränderungen eine bessere Fossilerhaltung ermöglichten. Während der Zeit nach der EW kehrte dies zu den Merkmalen vor der EW zurück (Erweiterte Daten, Abb. 8c und Abschnitt 11 mit ergänzenden Informationen).
Die stabilen Isotopendaten deuten darauf hin, dass δ13C und δ18O von G. blacki für die Zeit vor dem EW zwischen −16,2 und −13,8 ‰ bzw. −9,7 und −7,0 ‰ liegen. Während der EW steigt dieser leicht auf −15,3 bis −10,3 ‰ bzw. −9,3 bis −6,3 ‰. Im Fall von P. weidenreichi sind die δ13C- und δ18O-Bereiche vor EW ähnlich bei –14,7 bis –13,7‰ und –7,1 bis –6,3‰, erstrecken sich auf –14,7 bis –13,3‰ und ändern sich auf –4,9 und –4,4‰ während der EW-Periode (Abb. 3d, e, Erweiterte Daten Abb. 8b und Abschnitt 12 mit ergänzenden Informationen).
Die Spurenelementanalyse der G. blacki-Zähne vor der EW zeigt mehrere deutliche und synchrone Sr/Ca- und Ba/Ca-Streifen im Zahnschmelz und Dentin, die sich in deutlich weniger sichtbare diffuse Streifen näher an der EW ändern (Abb. 2d, erweitert). Daten Abb. 9 und 10a und ergänzende Informationen Abschnitt 13). Darüber hinaus ist in der Prä-EW eine deutliche Ableitungsstreifenbildung zu erkennen, die während der EW weniger deutlich wird (Extended Data Abb. 10a). Die Microwear-Analyse zeigt keine statistisch signifikanten Ernährungsunterschiede zwischen G. blacki- und P. weidenreichi-tragenden Standorten (Ergänzende Informationen, Abschnitt 14). Es gibt jedoch erhebliche Unterschiede in der Ernährung an vier G. blacki-tragenden Standorten zwischen der Zeit vor der EW und kurz vor der EW. G. blacki neigt dazu, etwas höhere Schwankungen der mittleren Anisotropie- und Komplexitätstrendlinien zu zeigen, wohingegen die von P. weidenreichi stabiler zu sein scheinen, insbesondere für die Anisotropie über die EW hinaus (Abb. 2c und 3f, g, erweiterte Daten Abb. 10b und). Ergänzende Informationen Abschnitt 14).
Zum ersten Mal wurde die größte Sammlung von In-situ-Beweisen von G. blacki über sein gesamtes Verbreitungsgebiet genau datiert, um eine genaue Zeitleiste für die Anwesenheit und Abwesenheit von G. blacki im Fossilienbestand zu liefern. Frühere Datierungen konzentrierten sich hauptsächlich auf die früheren Beweise für G. blacki2,8 und ortsspezifische Chronologien (z. B. Lit. 9). Im Gegensatz dazu wurde durch die Eingrenzung von Höhlen innerhalb des gesamten Altersbereichs sowohl im Chongzuo- als auch im Bubing-Becken ein genaueres regionales Fenster des Aussterbens bei 295–215 ka ermittelt.
Die Pollen- und Faunendaten deuten darauf hin, daß die frühen Mosaiklandschaften vor der EW in der Übergangsphase durch eine erhöhte Umweltvariabilität unterbrochen wurden (Abb. 3b), was durch die Veränderung der Waldgemeinschaften und -strukturen nahegelegt wird, und nach der EW, wie durch einen Rückgang der Baumbestände nahegelegt Bedeckung und eine Zunahme von Farnen und Grasland im Zusammenhang mit Bränden. Diese Variabilität begann schrittweise zwischen 1.100 und 350 ka und nahm ab etwa 200 ka dramatisch zu (Abb. 3b). Es wurde diese Variabilität als Verschiebungen hin zu erhöhter Saisonalität und trockeneren Umgebungen interpretiert, die zu einer Verlagerung hin zu saisonalen subtropischen/tropischen feuchten Tieflandwäldern und einer Zunahme von Sträuchern und offenen Graslandumgebungen vor und während der EW führten (Ergänzende Informationen, Abschnitt 10). Diese Umweltvariabilität zeigt sich auch in der Sedimentaufzeichnung, da die stabilen Niedrigenergieumgebungen der Vor-EW durch instabile Hochenergieumgebungen der EW ersetzt wurden, wobei die Wasserverfügbarkeit auf die Regenzeit beschränkt war (Erweiterte Daten, Abb. 8c und Abschnitt „Ergänzende Informationen“) 11).
Der Rückgang der Waldfläche in diesem Zeitraum ist in China21, Südostasien22 und Australasien23 dokumentiert. Die Pollenstudie zeigt jedoch, daß der Schlüssel zum Aussterben von G. blacki nicht in der Verschlechterung der Baumbedeckung liegt, sondern vielmehr im Einfluss von Umweltschwankungen auf die Veränderung der Zusammensetzung von Waldgemeinschaften, insbesondere in der Zunahme von Störtaxa. Die gewonnenen stabilen Isotopen- und Spurenelementdaten liefern neue Einblicke in das Ausmaß dieser Variabilität und die Auswirkungen auf G. blacki (Ergänzende Informationen, Abschnitte 12 und 13). Vor EW lebten G. blacki und P. weidenreichi beide in bewaldeten Umgebungen mit geschlossenen Baumkronen (Abb. 3b und erweiterte Daten, Abb. 8b) mit stärkerer biogener Streifenbildung (Abb. 2d(i)–(iii)), was wahrscheinlich auf a zurückzuführen ist größere Vielfalt an Nahrungsquellen, einschließlich saisonaler Früchte und Blumen und periodischer Wasserkonsum, wie durch die deutliche Bleistreifenmarkierung angezeigt (Erweiterte Daten Abb. 10a,b). Die wahrscheinlichsten Nahrungsquellen wären das ganze Jahr über in größerer Verfügbarkeit gewesen und hätten nur diskreten Stress in der Bevölkerung verursacht (Abb. 2d(i)–(iii)). Mit Ausnahme eines Individuums scheint G. blacki während der gesamten EW-Periode eine spezialisiertere geschlossene Baumkronennische unterhalten zu haben, die möglicherweise auf eine Mischung von Waldpflanzen angewiesen ist (Erweiterte Daten, Abb. 8b). Diese Spezialisierung während einer Umweltveränderung könnte zu einem diffuseren biogenen Signal im Zahngewebe der einzelnen Personen geführt haben (Abb. 2d(iv)–(v)), was auf eine stark verringerte Ernährungsvielfalt und einen geringeren regelmäßigen Wasserverbrauch schließen lässt (Erweiterte Daten, Abb. 10c). ,d) und erhöhter chronischer Stress in der Bevölkerung (Abb. 2d(iv)–(v)). Dies ist der erste Einblick in das Verhalten von G. blacki als einer Art am Rande des Aussterbens, was im krassen Gegensatz zu P. weidenreichi (Abb. 2d(vi)) steht, das zu diesem Zeitpunkt viel weniger Stress zeigt. Über den EW hinaus scheint sich P. weidenreichi verlagert zu haben, um die offeneren, saisonalen Lebensräume zu nutzen (Extended Data Abb. 8b) und möglicherweise weiterhin die saisonale Befruchtung von Früchten zu nutzen, wie es das moderne Pongo heute auf Borneo tut24.
Die Veränderungen der Mikroverschleißwerte bei G. blacki- und P. weidenreichi-Zähnen können auch mit Perioden der Fruchtknappheit zusammenhängen. G. blacki neigt dazu, spezifischere Ernährungspräferenzen zu zeigen (sowohl bei Früchten als auch bei faserhaltigen Nahrungsmitteln), was auf eine stärkere Abhängigkeit von faserhaltigen Ersatznahrungsmitteln hinweist (Abb. 2c), beispielsweise in der Ostwestküste, als das Klima saisonaler wurde und weniger Früchte verfügbar waren. Dies könnte G. blacki dazu gezwungen haben, seine Ernährung von ernährungsphysiologisch bevorzugten Komponenten in geringerem Angebot auf weniger nährstoffreiche Ersatznahrungsmittel in reichlichem Angebot umzustellen. Der erhöhte Verzehr von faserhaltigen Lebensmitteln bei P. weidenreichi im EW könnte auf eine bessere Umstellung auf Ersatznahrungsmittel und eine insgesamt flexiblere und ausgewogenere Ernährung hinweisen (Abb. 2c und erweiterte Daten, Abb. 10). Diese erste DMTA-Analyse des gesamten G. blacki-Materialspektrums bietet einen einzigartigen Einblick in seine Unfähigkeit, sich anzupassen, und seine möglicherweise schlechte Auswahl an Ersatznahrungsmitteln.
Diese Studie präsentiert einen genauen Zeitplan für das Vorkommen und Aussterben von G. blacki. In der Zeit vor der EW blühte G. blacki zusammen mit anderen Primaten als erfolgreicher Spezialist auf (Abb. 3c) und genoss eine große Nahrungsvielfalt in einem reichen immergrünen Laubwald (Abb. 2d(i)–(ii)) und reichlich Wasserquellen (Erweiterte Daten Abb. 10a–d) unter stabilen Umweltbedingungen (Abb. 2b). Etwa vor 700–600 ka kam es in der Übergangsphase zu einer Verschiebung hin zu einer zunehmenden Saisonalität, die zu einer Veränderung der Waldgemeinschaften (Abb. 3b), einer geringeren Vielfalt an Nahrungsquellen (Abb. 2d(iii)–(iv)) und instabilen hohen Temperaturen führte. Energieumgebungen (Erweiterte Daten, Abb. 8c), Veränderungen in der Zusammensetzung der Fauna und weit verbreitete Faunenumsätze (Abb. 3c und ergänzende Informationen, Abschnitt 2), eine Verlagerung hin zu saisonalen Lebensräumen durch P. weidenreichi (Erweiterte Daten, Abb. 8b) und a Veränderung der Ernährungsvielfalt und des Verhaltens von G. blacki (Abb. 2d und 3f, g).
Obwohl P. weidenreichi vor der EW eine ähnliche Umgebung hatte, gibt es zwischen 600 und 300 Jahren Hinweise darauf, daß G. blacki nicht in der Lage war, sich an diese Übergangszeit anzupassen, was einen größeren Einfluss auf seine Widerstandsfähigkeit gegenüber der sich verändernden Ökologie hatte. Die Abhängigkeit von G. blacki von Früchten und nährstoffärmeren Ersatznahrungsmitteln (Abb. 2c) führte zu einer risikoreicheren Futtersuchstrategie und machte ihn in Kombination mit seiner viel größeren, weniger beweglichen Körpergröße anfälliger für Veränderungen in der Waldstruktur25 (Abb . 2c). Darüber hinaus war G. blacki ausschließlich terrestrisch, möglicherweise mit einem kleinen geografischen Verbreitungsgebiet20, reiste jedoch regelmäßig das Tal hinunter, um Wasser zu verbrauchen (Erweiterte Daten Abb. 10a–d), wohingegen P. weidenreichi eher baumbewohnend, mobil und halbsolitär war und Wasser im Blatt sammelte Überdachung. Darüber hinaus deuten die einzigartigen dentognathen Merkmale13,14 und die riesige Körpergröße4,5 von G. blacki auf einen höheren Bedarf an Nahrungsaufnahme und ein langsameres und verzögerteres Wachstumsmuster hin, was auf eine geringere Reproduktionsrate schließen lässt26. Obwohl die Zahngröße von G. blacki im Laufe des Pleistozäns zunahm, was auch eine Zunahme der Körpergröße bedeutet, nahm die Größe von P. weidenreichi ab27. Dies macht ihn zu einem agileren Adapter. P. weidenreichi zeigte auch eine Flexibilität gegenüber offenen Lebensräumen (Extended Data Abb. 8b), die sich möglicherweise in kleineren Gruppen bewegten, und war in der Lage, sein Verhalten als Reaktion auf Umweltschwankungen anzupassen, was zu einer weniger gestressten Population führte (Abb. 3d).
Vor etwa 300 Jahren gibt es Hinweise auf eine kämpfende G. blacki-Population, da die Anzahl der G. blacki-Höhlen und -Zähne abnahm (Abb. 3c), was auf eine schrumpfende Population hindeutet. Die starke Veränderung der Zahnbänder von G. blacki weist auf chronischen Stress in der Bevölkerung hin (Abb. 2d(iv)–(v)) und weist auf Veränderungen des bevorzugten Ernährungsverhaltens (Abb. 2c und Extended Data Abb. 10f, g) hin dass G. blacki Schwierigkeiten hatte, auf die Umweltveränderungen in einem potenziell schrumpfenden Gebiet zu reagieren20. Es scheint, daß seine Waldrefugien ihre Struktur verändert haben und zu offen und gestört geworden sind, als dass sich diese Art ernähren könnte. Im Vergleich zu anderen bekannten Aussterbeereignissen in Nordamerika und Australien, die vom Homo sapiens28,29,30 beeinflusst wurden, gibt es keine Hinweise darauf, dass archaische Homininen bei diesem früheren Aussterben der Megafauna in Südchina eine Rolle gespielt haben.
Die Angabe einer definierten Ursache für das Aussterben ist für viele ausgestorbene Arten eine Leistung, die selten erreicht wurde, da sie einen gattungs- und artspezifischen Ansatz erfordert28. Obwohl es sehr schwierig sein kann, die genauen Ursachen für die Ausrottung und das Aussterben der Megafauna zu bestimmen29,30, liefert unsere Multiproxy-Aufzeichnung des Zeitpunkts, der Umwelt und des Verhaltens von G. blacki fundierte regionale Einblicke in den ökologischen Kontext dieser Art. G. blacki war der ultimative Spezialist und als sich die Baumumgebung veränderte, besiegelte sein Kampf um die Anpassung sein Schicksal. Im Vergleich dazu verbreitete und diversifizierte sich der generalistische Homo in dieser Zeit über ganz Südostasien und schien die neuen Mosaikumgebungen, die für G. blacki ein solches Problem darstellten, flexibel ausgenutzt zu haben. Insgesamt liefert unser Datensatz einen wichtigen Kontext für die sich verändernden Schicksale verschiedener Primatenarten in Südostasien und wirft neues Licht auf den Untergang des größten Primaten, der jemals auf dem Planeten gelebt hat.